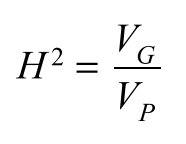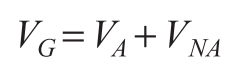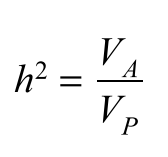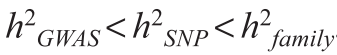本文关键词: Heritability, h2, family heritability , SNP heritability, GWAS heritability , Missing heritability
遗传力的基础概念
遗传力(Heritability)是我们理解遗传与环境因素对性状影响的基础,定义为遗传方差占性状方差(总方差)的比值,可以理解为遗传因素对性状的影响,数学上以h2表示。
通常根据对遗传方差的定义而分为广义与狭义遗传力:
广义遗传力 broad-sense heritability :
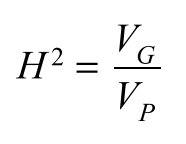 其中VP = VG + VE, VP为性状方差,VE为环境方差(也包括测量误差等),而分子的VG为遗传方差。
其中VP = VG + VE, VP为性状方差,VE为环境方差(也包括测量误差等),而分子的VG为遗传方差。
遗传方差VG也可进一步细分为
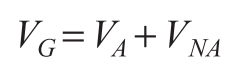 VA是加性遗传效应的方差,VNA是非加性遗传效应的方差 (上位与显性遗传效应)
VA是加性遗传效应的方差,VNA是非加性遗传效应的方差 (上位与显性遗传效应)
加性遗传效应是指当两个或多个基因对于某一性状,或是单个基因的不同等位基因对于某一性状的整体作用,等于它们单独作用之和。
非加性遗传效应 则包括 上位与显性遗传效应 。
因为对于绝大多数复杂性状,很少有证据证明有 非加性遗传效应存在,所以我们目前聚焦于主要考虑 加性遗传效应 的 狭义遗传力,
狭义遗传力 narrow-sense heritability :
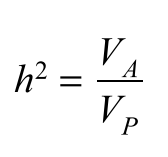
遗传力的估计
我们有多种方法可以估计遗传力h2的大小,目前主要的方法有三种,通过双胞胎研究,SNP或是GWAS来估计。
h2 family : 双胞胎研究,通过比较同卵与异卵双胞胎的相似性,计算得到h2,通常为这三种中最高。
h2 SNP :GWAS研究所用chip上所有variants共同解释的方差 与 性状方差的比值,比 h2 family 低,但会显著高于h2 GWAS。可以使用GCTA的GREML模型来估计。(使用GCTA (GREML)来估计SNP-遗传力 SNP Heritability )
h2 GWAS :仅由GWAS所发现的某疾病相关variants解释的方差 与 性状方差的比值 ,三者中最低。
一般情况下,三者的关系:
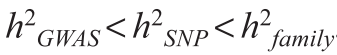
更直观的关系如下图所示:
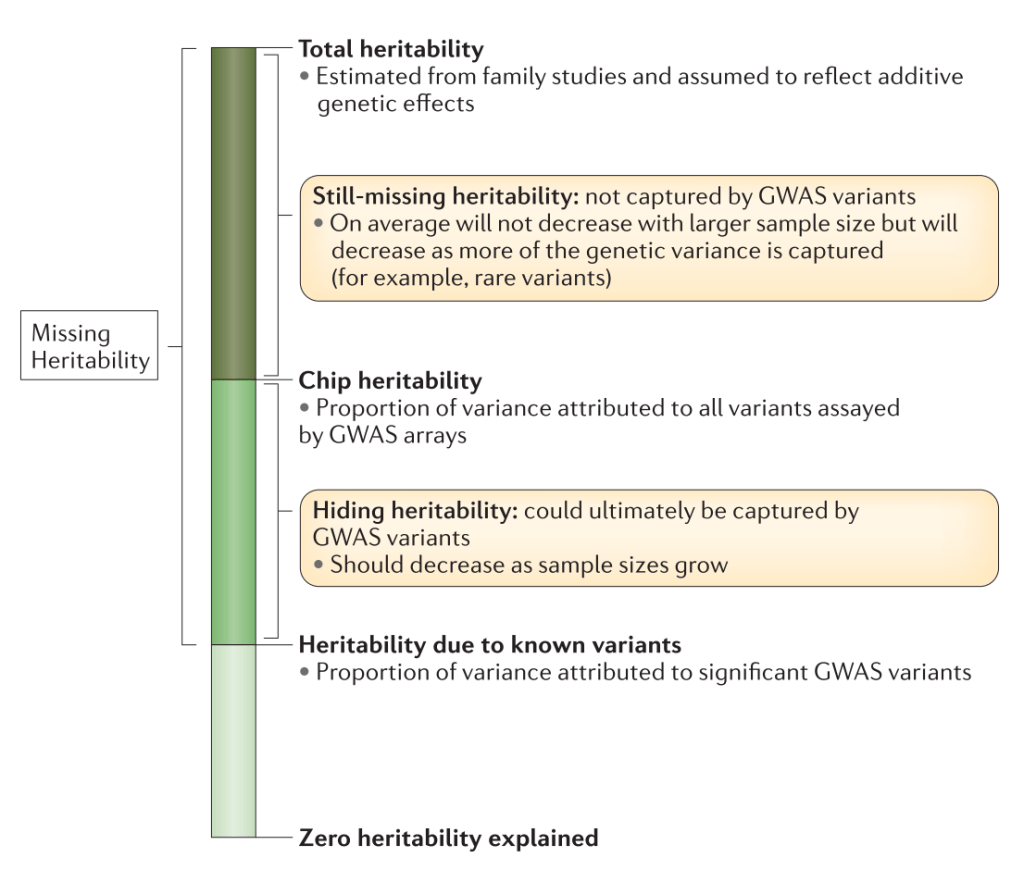 图一:三种遗传力的关系。(引自:The contribution of genetic variants to disease depends on the ruler,2014)
图一:三种遗传力的关系。(引自:The contribution of genetic variants to disease depends on the ruler,2014)
Missing and hidden heritability
GWAS研究中一个核心问题便是 Missing heritability, 定义为 h2 family 与 h2 GWAS 之间的差值。 h2 GWAS 之所以低于 h2 family ,潜在的原因包括:非加性遗传效应(尽管目前证据很少),效应量大的稀有变异(rare variants),或是双胞胎研究中由于共同的环境因素而造成的过高估计。
Missing heritability 又可细分为 still- missing heritability 与 hidden heritability 。 still- missing heritability 为 h2 family 与 h2 SNP 之间的差,Yang 认为可能的原因是在GWAS研究中由于样本数量的限制,大多数效应量较小的遗传效应无法被可靠地检测。
而 hidden heritability 则为 h2 SNP 与 h2 GWAS 的差。对它的理解建立在Fisher最初对于无穷小模型(infinitesimal model),即多数变异都只有很小的效应。在GWAS研究中,由于我们所选显著阈值的高低,遗传力或许并不是 消失( missing ) 而是被隐藏( hidden )了。另一种可能则是,人群的异质性(heterogeneity.),因为 h2 GWAS 大多来自包含多群体的meta分析,而遗传效应在这些群体中的异质性也可能使 h2 GWAS 偏低。
如何估计SNP遗传力:
使用GCTA (GREML)来估计SNP-遗传力 SNP Heritability
参考:
An Introduction to Statistical Genetic Data Analysis.